Molecular evolution of influenza A/H3N2 viruses HA and NA genes circulating in Shenzhen in 2017
-
摘要:
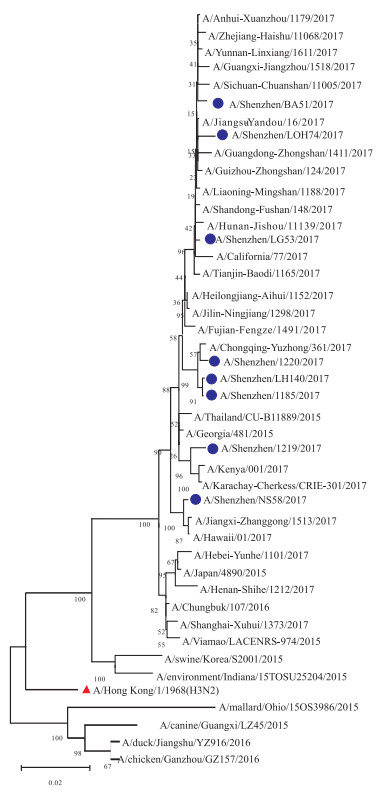
目的 了解深圳市2017年上半年流行的甲型H3N2亚型流感病毒HA和NA基因的分子进化特征,为预测流感病毒流行和变异提供科学依据。 方法 利用DNAStar、MEGA 7.0等生物信息学软件对研究分离的40株H3N2流感病毒的HA和NA基因及其编码的氨基酸序列进行比对,构建分子进化树,分析基因特征和变异情况。 结果 深圳分离株的同源性均达到97.8%~100.0%,位于亚洲北美人源分支。与世界卫生组织(World Health Organization,WHO)推荐的疫苗株A/Switzerland/9715293/2013(H3N2)和A/Hong Kong/4801/2014(H3N2)相比,有较高的序列相似性;与当年疫苗株对比发现,HA发生6个抗原位点和2个受体结合位点的改变,NA出现第151位酶活性位点的改变;HA和NA的潜在N-糖基化位点在数量和位置上也发生变化。 结论 深圳市2017年上半年甲型H3N2亚型流感病毒在流行过程中尚未发生明显变异,目前推荐的疫苗株仍对深圳地区人群有一定保护作用,HA和NA多个氨基酸位点的变异提示仍需加强H3N2亚型流感病毒分子水平的动态监测。 -
关键词:
- H3N2亚型流感病毒 /
- HA基因 /
- NA基因 /
- 分子进化 /
- 序列分析
Abstract:Objective To investigate the molecular evolution of the Hemagglutinin (HA) and Neuraminidase (NA) genes of influenza A/H3N2 viruses in Shenzhen in the first half of 2017, so as to provide scientific basis for predicating influenza epidemic and variation. Methods A total of 40 influenza A/H3N2 viruses strains were selected and the molecular phylogenetic trees were constructed by bioinformatics software DNAStar, MEGA 7.0, etc. Then, the genetic characteristics and variation of HA and NA genes along with corresponding amino acids were analyzed. Results The homology of Shenzhen isolates reached 97.8%-100.0%, which located in the human-derived branch of Asia and North America. Compared with the vaccine strains A/Switzerland/9715293/2013(H3N2) and A/Hong Kong 14801/2014(H3N2) recommended by world Health Oraganication (WHO), there was a higher sequence similarity. Compared with the vaccine strain, HA and NA proteins had a number of amino acid sites replaced, of which HA 6 antigen sites and 2 receptor binding sites change; NA had a mutation of D151N/G located in enzyme activity sites. Potential N-glycosylation sites for HA and NA also changed. Conclusions The influenza A/H3N2 viruses in Shenzhen in the first half of 2017 has not yet formed a new subtype in the epidemic. Currently, the recommended vaccine strains still have some protective effects on the population. The replacement mutation of multiple amino acids sites of HA and NA suggests that the dynamic monitoring of molecular level of influenza A/H3N2 viruses need to be strengthened. -
Key words:
- H3N2 subtype influenza virus /
- HA gene /
- NA gene /
- Molecular evolution /
- Sequence analysis
-
表 1 35株国内外近3年H3N2流感病毒参考株
Table 1. 35 domestic and overseas reference strains of H3N2 IAV in recently 3 years
谱系 地区(州) 国家 H3N2流感病毒疫苗株名称 GenBank/GISAID登记号 HA NA 人类 亚洲 中国 A/Guangdong-Zhongshan/1411/2017 EPI1029982 EPI1029981 人类 亚洲 中国 A/Anhui-Xuanzhou/1179/2017 EPI1030195 EPI1030194 人类 亚洲 中国 A/Hebei-Yunhe/1101/2017 EPI1030198 EPI1030197 人类 亚洲 中国 A/Tianjin-Baodi/1165/2017 EPI1030270 EPI1030269 人类 亚洲 中国 A/Liaoning-Mingshan/1188/2017 EPI1030273 EPI1030272 人类 亚洲 中国 A/Guizhou-Zhongshan/124/2017 EPI1030336 EPI1030335 人类 亚洲 中国 A/Henan-Shihe/1212/2017 EPI1030345 EPI1030344 人类 亚洲 中国 A/Heilongjiang-Aihui/1152/2017 EPI1030348 EPI1030347 人类 亚洲 中国 A/Sichuan-Chuanshan/11005/2017 EPI1062397 EPI1062396 人类 亚洲 中国 A/Jiangxi-Zhanggong/1513/2017 EPI1062595 EPI1062594 人类 亚洲 中国 A/Zhejiang-Haishu/11068/2017 EPI1062721 EPI1062720 人类 亚洲 中国 A/Fujian-Fengze/1491/2017 EPI1062727 EPI1062726 人类 亚洲 中国 A/Jiangsu-Yandou/16/2017 EPI1062805 EPI1062804 人类 亚洲 中国 A/Hunan-Jishou/11139/2017 EPI1062898 EPI1062897 人类 亚洲 中国 A/Yunnan-Linxiang/1611/2017 EPI1062901 EPI1062900 人类 亚洲 中国 A/Chongqing-Yuzhong/361/2017 EPI1062907 EPI1062906 人类 亚洲 中国 A/Shanghai-Xuhui/1373/2017 EPI1062934 EPI1062933 人类 亚洲 中国 A/Jilin-Ningjiang/1298/2017 EPI1062955 EPI1062954 人类 亚洲 中国 A/Guangxi-Jiangzhou/1518/2017 EPI1062964 EPI1062963 人类 亚洲 中国 A/Shandong-Fushan/148/2017 EPI1067694 EPI1067739 人类 亚洲 泰国 A/Thailand/CU-B11889/2015 KU558953 KX151205 人类 亚洲 韩国 A/Chungbuk/107/2016 KY509835 KY509841 人类 亚洲 日本 A/Japan/4890/2015 CY208123 CY209917 人类 欧洲 俄罗斯 A/Karachay-Cherkess/CRIE-301/2017 MF736999 MF737001 人类 北美洲 美国 A/Hawaii/01/2017 CY230946 CY230948 人类 北美洲 美国 A/California/77/2017 CY245366 CY245368 人类 非洲 肯尼亚 A/Kenya/001/2017 MF599417 MF599445 人类 南美洲 巴西 A/Viamao/LACENRS-974/2015 KY925971 KY926260 人类 南美洲 格鲁吉亚 A/Georgia/481/2015 KT252564 KT252565 猪 亚洲 韩国 A/swine/Korea/S2001/2015 KT427456 KT427458 禽类 亚洲 中国 A/duck/Jiangshu/YZ916/2016 MG021165 MG021167 禽类 亚洲 中国 A/chicken/Ganzhou/GZ157/2016 KY415634 KY415722 禽类 北美洲 美国 A/mallard/Ohio/15OS3986/2015 KY583991 KY584037 犬类 亚洲 中国 A/canine/Guangxi/LZ45/2015 MG254069 MG254100 环境 北美洲 美国 A/environment/Indiana/15TOSU25204/2015 KY327506 KY327751 表 2 8株代表株HA氨基酸序列主要的N-糖基化位点
Table 2. N-Glycosylation sites of HA amino acid sequence
代表株名称 潜在N-糖基化位点 24 38 61 79 138 149 151 180 181 300 301 498 A/Hong Kong/1/1968 NST NGT - - - - - NVT - NGS - NGT A/Hong Kong/4801/2014 NST NGT - NCT - NGT - - NVT - NGS - A/Shenzhen/LH140/2017 NST NGT - NCT NES - NSS - NVT - NGS - A/Shenzhen/1185/2017 NST NGT - NCT NES - NSS - NVT - NGS - A/Shenzhen/BA51/2017 NST NGT - NCT - NGT - - NVT - NGS - A/Shenzhen/1220/2017 NST NGT - NCT NES NGT - - NVT - NGS - A/Shenzhen/NS58/2017 NST NGT - NCT - NGT - - NVT - NGS - A/Shenzhen/1219/2017 NST NGT - NCT NES - - - NVT - NGS - A/Shenzhen/LG53/2017 NST NGT - NCT - NGT - - NVT - NGS - A/Shenzhen/LOH74/2017 NST NGT NST NCT - NGT - - NVT - NGS - 注:“NST”、“NGT”、“NCT”、“NES”、“NSS”、“NVT”、“NGS”分别表示具体的糖基化形式,字母为氨基酸的缩写;“-”表示不存在此糖基化位点。 表 3 8株代表株NA氨基酸序列主要的N-糖基化位点
Table 3. N-Glycosylation sites of NA amino acid sequence
代表株名称 潜在N-糖基化位点 61 146 151 234 329 402 A/Hong Kong/1/1968 NIT NDT - NGT - NRS A/Hong Kong/4801/2014 NIT NNT - NGT - - A/Shenzhen/1152/2017 NIT - - NGT NDS - A/Shenzhen/1164/2017 NIT - - NGT - - A/Shenzhen/1220/2017 NIT - - NGT - - A/Shenzhen/BA50/2017 NIT NNT - NGT - - A/Shenzhen/BA52/2017 NIT - NRT NGT - - A/Shenzhen/LOH75/2017 NIT - - NGT - - A/Shenzhen/NS73/2017 NIT - - NGT - - A/Shenzhen/NS58/2017 NIT - - NGT - - 注:“NIT”、“NDT”、“NNT”、“NRT”、“NGT”、“NDS”、“NRS”分别表示具体的糖基化形式,字母为氨基酸的缩写;“-”表示不存在此糖基化位点。 -
[1] Kim H, Webster RG, Webby RJ. Influenza virus: dealing with a drifting and shifting pathogen[J]. Viral Immunol, 2018, 31(2): 174-183. DOI: 10.1089/vim.2017.0141. [2] Su S, Fu XL, Li GR, et al. Novel influenza D virus: epidemiology, pathology, evolution and biological characteristics[J]. Virulence, 2017, 8(8): 1580-1591. DOI: 10.1080/21505594.2017.1365216. [3] van de Wakker SI, Fischer MJE, Oosting RS. New drug-strategies to tackle viral-host interactions for the treatment of influenza virus infections[J]. Eur J Pharmacol, 2017, 809: 178-190. DOI: 10.1016/j.ejphar.2017.05.038. [4] Webster RG, Bean WJ, Gorman OT, et al. Evolution and ecology of influenza A viruses[J]. Microbiol Immunol, 1992, 56(1): 152-179. DOI: 10.1007/82_2014_396. [5] Shao WH, Li XX, Goraya MU, et al. Evolution of influenza A virus by mutation and re-assortment[J]. Int J Mol Sci, 2017, 18(8): 1650. DOI: 10.3390/ijms18081650. [6] Palese P, Shaw ML. Orthomyxoviridae: the viruses and their replication[M]. Philadelphia: Japan Virus Society, 2007. [7] Nelson MI, Viboud C, Simonsen L, et al. Multiple reassortment events in the evolutionary history of H1N1 influenza A virus since 1918[J]. PLoS Pathog, 2008, 4(2): e1000012. DOI: 10.1371/journal.ppat.1000012. [8] 陈国清, 李春香, 邵荣标, 等. 2015年盐城市甲型H3N2亚型流感病毒HA1基因分子进化特征分析[J]. 江苏预防医学, 2017, 28(2): 137-140. DOI: 10.13668/j.issn.1006-9070.2017.02.05.Chen GQ, Li CX, Shao RB, et al. Analysis of the molecular evolutional characteristics of the HA1 gene of the influenza A H3N2 virus isolated in Yancheng city in 2015[J]. Jiangsu J Prevent Med, 2017, 28(2): 137-140. DOI: 10.13668/j.issn.1006-9070.2017.02.05. [9] 深圳新闻网. 深圳流感活跃水平呈下降趋势, 未报告流感死亡病例[EB/OL]. (2017-08-07)[2017-09-17]. http://www.sznews.com/news/content/2017-08/07/content_16938853.htm.Shenzhen News Network. The level of flu activity in Shenzhen showed a downward trend, and no deaths from influenza were reported[EB/OL]. (2017-08-07)[2017-09-17]. http://www.sznews.com/news/content/2017-08/07/content_16938853.htm. [10] 深圳市疾病预防控制中心. 深圳市每周流感指数[EB/OL]. (2017-01-01)[2017-09-17]. http://www.szcdc.net/rdzt/lgzs/mzlgzs/.Shenzhen Center for Disease Control and Prevention. Weekly influenza index in Shenzhen[EB/OL]. (2017-01-01)[2017-09-17]. http://www.szcdc.net/rdzt/lgzs/mzlgzs/. [11] 李希妍, 成艳辉, 谭敏菊, 等. 2013-2014年度中国H3N2亚型流感病毒病原学特征分析[J]. 病毒学报, 2015, 31: 30-35. DOI: 10.13242/j.cnki.bingduxuebao.002622.Li XY, Cheng YH, Tan MJ, et al. Virological characteristics of influenza A (H3N2) virus in mainland China during 2013-2014[J]. Chin J Virol, 2015, 31: 30-35. DOI: 10.13242/j.cnki.bingduxuebao.002622. [12] World Health Organization. Recommended composition of influenza virus vaccines for use in the 2015-2016 northern hemisphere influenza season[J]. Weekly Epidemiological Record. 2015, 90(11): 97-108. DOI:http://dx.doi.org/. [13] World Health Organization. Recommended composition of influenza virus vaccines for use in the 2017-2018 northern hemisphere influenza season[J]. Weekly Epidemiological Record. 2017, 92(11): 117-128. http://www.ncbi.nlm.nih.gov/pubmed/25771542 [14] Gupta R, Jung E, Brunak S. Prediction of N-glycosylation sites in human proteins, 2004. [15] Palese P, Tobita K, Ueda M, et al. Characterization of temperature-sensitive influenza virus mutants defective in neuramindiase[J]. Virology, 1974, 61: 397-410. DOI: 10.1063/1.4807511. [16] 张晶波, 张家淮, 胡晓芬, 等. 北京市西城区2009-2011年H3N2甲型流感病毒HA1基因特性分析[J]. 中国卫生检验杂志, 2013, 23(3): 143-145. https://www.cnki.com.cn/Article/CJFDTOTAL-ZWJZ201303044.htmZhang JB, Zhang JH, Hu XF, et al. Gene analysis of HA1 of A influenza virus H3N2 in Xicheng district, Beijing[J]. Chin J Hea Lab Tech, 2013, 23(3): 143-145. https://www.cnki.com.cn/Article/CJFDTOTAL-ZWJZ201303044.htm [17] Wiley DC, Wilson IA, Skehel JJ, et al. Structural dentification of the antibody-binding sites of Hong Kong influenza hemagglutinin and their involvement in antigenic variation[J]. Nature, 1981, 289: 373-378. DOI: 10.1038/289373a0. [18] Weis W, Brown JH, Cusack S, et al. Structure of the influenza virus hemagglutinin complexed with its receptor, sialic acid[J]. Nature, 1998, 333: 426-431. DOI: 10.1038/333426a0. [19] Yen HL, Hoffmann E, Taylor G, et al. Importance of neuraminidase active-site residues to the neuraminidase inhibitor resistance of influenza viruses[J]. Virology, 2006, 80(17): 8787-8795. DOI: 10.1128/JVI.00477-06. [20] Zurcher T, Yates PJ, Daly J, et al. Mutations conferring zanamivir resistance in human influenza virus N2 neuraminidases compromise virus fitness and are not stably maintained invitro[J]. J Antimicrob Chemother, 2006, 58(4): 723-73. DOI: 10.1093/jac/dkl321. [21] Lin YP, Gregory V, Collins P, et al. Neuraminidase receptor binding variants of human influenza A(H3N2) viruses resulting from substitution of aspartic acid 151 in the catalytic site: a role in virus attachment?[J]. Virology, 2010, 84(13). DOI: 10.1128/JVI.00458-10. [22] Sun SS, Wang QZ, Zhao F, et al. Prediction of biological functions on glycosylation site migrations in human influenza H1N1 viruses[J]. PLoS One, 2012, 7(2): e32119. DOI: 10.1371/journal.pone.0032119. [23] Schweiger B, Zadow I, Heckler R. Antigenic drift and variability of influenza viruses[J]. Med Microbiol Immunol, 2002, 191(3): 133-138. DOI: 10.1007/s00430-002-0132-3. -




 下载:
下载: