Prognostic study of mild cognitive impairment progressing to Alzheimer′s disease based on polygenic risk score and machine learning modeling strategy
-
摘要:
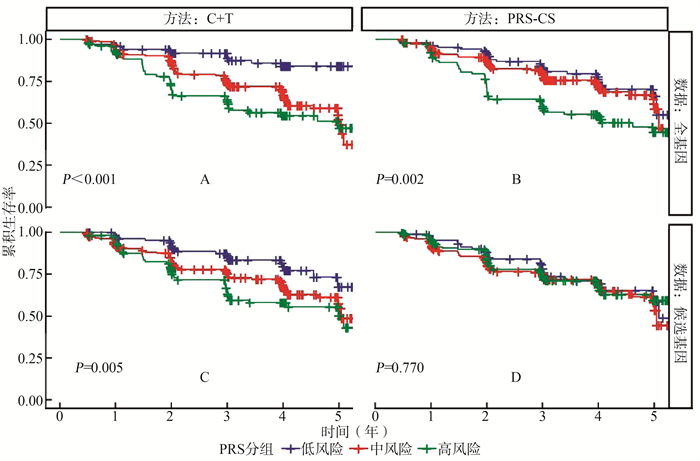
目的 本研究从全基因组和候选基因组的角度探究多基因风险评分(polygenic risk score, PRS)与机器学习对轻度认知障碍(mild cognitive impairment, MCI)发展为阿尔茨海默病(alzheimer′s disease, AD)的预后预测性能, 为MCI发展为AD的第5年预后预测建模提供更有力的方法理论依据。 方法 借助聚类与阈值(clumping and thresholding, C+T)、多基因风险评分-连续收缩(polygenic risk scores-continuous shrinkage, PRS-CS)、随机生存森林(random survival forest, RSF)、生存支持向量机(survival support vector machine, SSVM)4种常用统计方法对MCI发展为AD的第5年生存情况进行预测建模。利用C+T与PRS-CS得到的AD遗传风险得分作为独立的预测因子纳入Cox比例风险回归模型, RSF与SSVM则从候选基因组角度直接纳入所有与AD有关的单核苷酸多态性(single nucleotide polymorphism, SNPs)进行统计建模。最后, 采用C指数作为模型预测效果的评价指标。 结果 无论是C+T还是PRS-CS方法, 在全基因组和候选基因组两种情况下的C指数差值均 < 0.01, 而两种方法的C指数差值最大为0.04, 二者差异均无统计学意义; 机器学习的方法明显好于PRS方法, RSF和SSVM的C指数均能达到0.76, 较C+T、PRS-CS高0.07、0.11, 差异有统计学意义(均P < 0.05)。 结论 机器学习方法表现优异, 为MCI发展为AD的预后预测提供了更为可行的统计建模方案。 Abstract:Objective To provide the theoretical basis for modeling the fifth-year prognostic prediction of the conversion from mild cognitive impairment (MCI) to alzheimer′s disease (AD), this study explored the prognostic prediction performance of polygenic risk score and machine learning methods on the progression from MCI to AD from the perspective of whole genome and candidate genome. Methods Using clumping and thresholding (C+T), polygenic risk scores-continuous shrinkage (PRS-CS), random survival forest (RSF), and survival support vector machine (SSVM) to predict the fifth-year prognostic prediction of patients who progressed from MCI to AD.The polygenic risk score of AD obtained by C+T and PRS-CS were included as independent predictors in Cox proportional hazards regression model, while RSF and SSVM were directly included in all single nucleotide polymorphisms (SNPs) related to AD from the perspective of candidate genome for statistical modeling.Finally, C-index was used as the evaluation index of the prediction effect of the model. Results The difference in C-index between the whole genome and candidate genome was less than 0.01 for both C+T and PRS-CS methods, while the maximum difference of C-index between the two methods was 0.04, and there was no statistical difference between them.The machine learning methods significantly outperformed the PRS methods.The C-index of RSF and SSVM reached 0.76, indicating significance increases of 0.07 and 0.11 over C+T and PRS-CS, respectively (all P < 0.05). Conclusions Machine learning methods perform well and provide a more feasible statistical modeling scheme for the prognostic prediction of the progression of MCI to AD. -
表 1 基本信息
Table 1. Basic information
变量 MCI(n=299) AD(n=130) 年龄/岁(x±s) 72.57±7.39 73.46±6.85 受教育程度(x±s) 15.97±2.87 16.09±2.91 性别[人数(占比/%)] 男 181(60.53) 81(62.30) 女 118(39.47) 49(37.70) 注:1. MCI:轻度认知障碍。2. AD:阿尔茨海默病。 表 2 PRS方法与机器学习方法十折交叉验证结果均值的多重比较
Table 2. Multiple comparisons of the means of ten-fold cross-validation results between the PRS method and machine learning method
方法Ⅰ 方法Ⅱ 95% CI P值 PRS-CS C+T -0.098 1~0.020 2 0.190 RSF -0.167 2~-0.048 9 0.001 SSVM -0.169 2~-0.050 9 0.001 C+T PRS-CS -0.020 2~0.098 1 0.190 RSF -0.128 3~-0.010 0 0.023 SSVM -0.130 3~-0.012 0 0.020 RSF PRS-CS 0.048 9~0.167 2 0.001 C+T 0.010 0~0.128 3 0.023 SSVM -0.061 2~0.057 2 0.946 SSVM PRS-CS 0.050 9~0.169 2 0.001 C+T 0.012 0~0.130 3 0.020 RSF -0.057 2~0.061 2 0.946 注:1. RSF:随机生存森林。2. PRS-CS:多基因风险评分-连续收缩。3. C+T: 聚类与阈值。4. SSVM:生存支持向量机。5. 多重比较的P值使用Bonferroni法进行校正。 -
[1] Nussbaum RL, Ellis CE. Alzheimer's disease and parkinson's disease[J]. N Engl J Med, 2003, 348(14): 1356-1364. DOI: 10.1056/NEJM2003ra020003. [2] Petersen RC. Mild cognitive impairment[J]. Continuum (Minneap Minn), 2016, 22(2 Dementia): 404-418. DOI: 10.1212/CON.0000000000000313. [3] Li HT, Yuan SX, Wu JS, et al. Predicting conversion from MCI to AD combining multi-modality data and based on molecular subtype[J]. Brain Sci, 2021, 11(6): 674. DOI: 10.3390/brainsci11060674. [4] Mamani NM. Machine learning techniques and polygenic risk score application to prediction genetic diseases[J]. ADCAIJ, 2020, 9(1): 5-14. DOI: 10.14201/ADCAIJ20209154. [5] Baker E, Escott-Price V. Polygenic risk scores in Alzheimer's disease: current applications and future directions[J]. Front Digit Health, 2020, 2: 14. DOI: 10.3389/fdgth.2020.00014. [6] Li J, Lu Q, Wen Y. Multi-kernel linear mixed model with adaptive lasso for prediction analysis on high-dimensional multi-omics data[J]. Bioinformatics, 2020, 36(6): 1785-1794. DOI: 10.1093/bioinformatics/btz822. [7] Privé F, Vilhjálmsson BJ, Aschard H, et al. Making the most of clumping and thresholding for polygenic scores[J]. Am J Hum Genet, 2019, 105(6): 1213-1221. DOI: 10.1016/j.ajhg.2019.11.001. [8] Ge T, Chen CY, Ni Y, et al. Polygenic prediction via Bayesian regression and continuous shrinkage priors[J]. Nat Commun, 2019, 10(1): 1776. DOI: 10.1038/s41467-019-09718-5. [9] Ishwaran H, Kogalur UB, Blackstone EH, et al. Random survival forests[J]. Thorac Oncol, 2008, 2(3): 841-860. DOI: 10.1214/08-AOAS169. [10] Evers L, Messow CM. Sparse kernel methods for high-dimensional survival data[J]. Bioinformatics, 2008, 24(14): 1632-1638. DOI: 10.1093/bioinformatics/btn253. [11] Deng Y, Cheng S, Huang H, et al. Toward better risk stratification for implantable cardioverter-defibrillator recipients: implication of machine learning models[J]. J Cardiovasc Dev Dis, 2022, 9(9): 310-325. DOI: 10.3390/jcdd9090310. [12] Kunkle BW, Grenier-Boley B, Sims R, et al. Genetic meta-analysis of diagnosed Alzheimer's disease identifies new risk loci and implicates Aβ, tau, immunity and lipid processing[J]. Nat Genet, 2019, 51(3): 414-430. DOI: 10.1038/s41588-019-0358-2. [13] Leonenko G, Baker E, Stevenson-Hoare J, et al. Identifying individuals with high risk of Alzheimer's disease using polygenic risk scores[J]. Nat Commun, 2021, 12(1): 4506. DOI: 10.1038/s41467-021-24082-z. [14] Ritchie K. Mild cognitive impairment: an epidemiological perspective[J]. Dialogues Clin Neurosci, 2004, 6(4): 401-408. DOI: 10.31887/DCNS.2004.6.4/kritchie. -




 下载:
下载: